机器学习驱动的分子嵌入助力预测臭氧氧化去除新兴污染物
在饮用水处理过程中,臭氧在去除新出现的污染物(ECs)方面具有很高的功效。然而,传统的定量结构激活关系(QSAR)模型往往不能有效地规范化和表征不同的分子结构,从而限制了其对各种ec去除的预测准确性。本研究使用由图神经网络(GNN)生成的嵌入式分子结构向量,结合功能群提示,作为前馈神经网络的输入。建立了28个ec和542个数据点的数据集,代表了不同的分子结构和物理化学性质,以预测ec (REC)在臭氧氧化中的残留率。与传统的QSAR模型相比,基于gnn的分子结构嵌入方法显著提高了预测精度。由此产生的KANO-EC模型REC的R2为0.97,证明了其捕获复杂结构特征的能力。此外,KANO-EC保持了出色的可解释性,阐明了参与氧化机制的关键官能团(如羰基、羟基、芳香环和胺)。本研究提出了KANO-EC模型作为预测电流和电位ec的臭氧氧化去除效率的新方法。该模型还为制定有效的控制战略以确保饮用水供应的长期安全和可持续性提供了宝贵的见解。
主要研究发现
本研究引入GNN生成的分子结构嵌入向量,并结合功能团提示,作为前馈神经网络的输入。基于28种ECs和542组实验数据,构建了涵盖多种分子结构与理化性质的数据集,用于预测臭氧氧化后的REC。与传统QSAR模型相比,GNN嵌入方法显著提升了预测准确性。所构建的KANO–EC模型在REC预测中实现了R²值高达0.97,展现出对复杂结构特征的精准捕捉能力。同时,该模型保持了良好的可解释性,成功识别出羰基、羟基、芳环、胺等关键功能团在臭氧反应机制中的作用。本研究提出的KANO–EC模型为评估现有及潜在ECs的臭氧去除效率提供了新方法,并为保障饮用水系统的长期安全与可持续运行提供了理论支持。
本研究探索了四种分子结构嵌入路径对模型性能与泛化能力的影响(图1)。首先采用ECFP提取基于环状拓扑的结构指纹,生成1024位分子指纹表征分子活性特征(图1a)。其次,采用SMILES对编码(SMILES Pair Encoding,SPE)方法构建SMILES子串词汇表并编码为向量,适用于QSAR建模但可解释性较弱(图1b)。D-MPNN利用定向边传播机制更新原子与键的特征信息,生成嵌入向量以捕捉局部结构环境(图1c)。KANO模型则基于知识图谱识别功能团语义,通过注意机制生成功能提示并融合至原子表示中,提升结构表达与预测性能(图1d)。
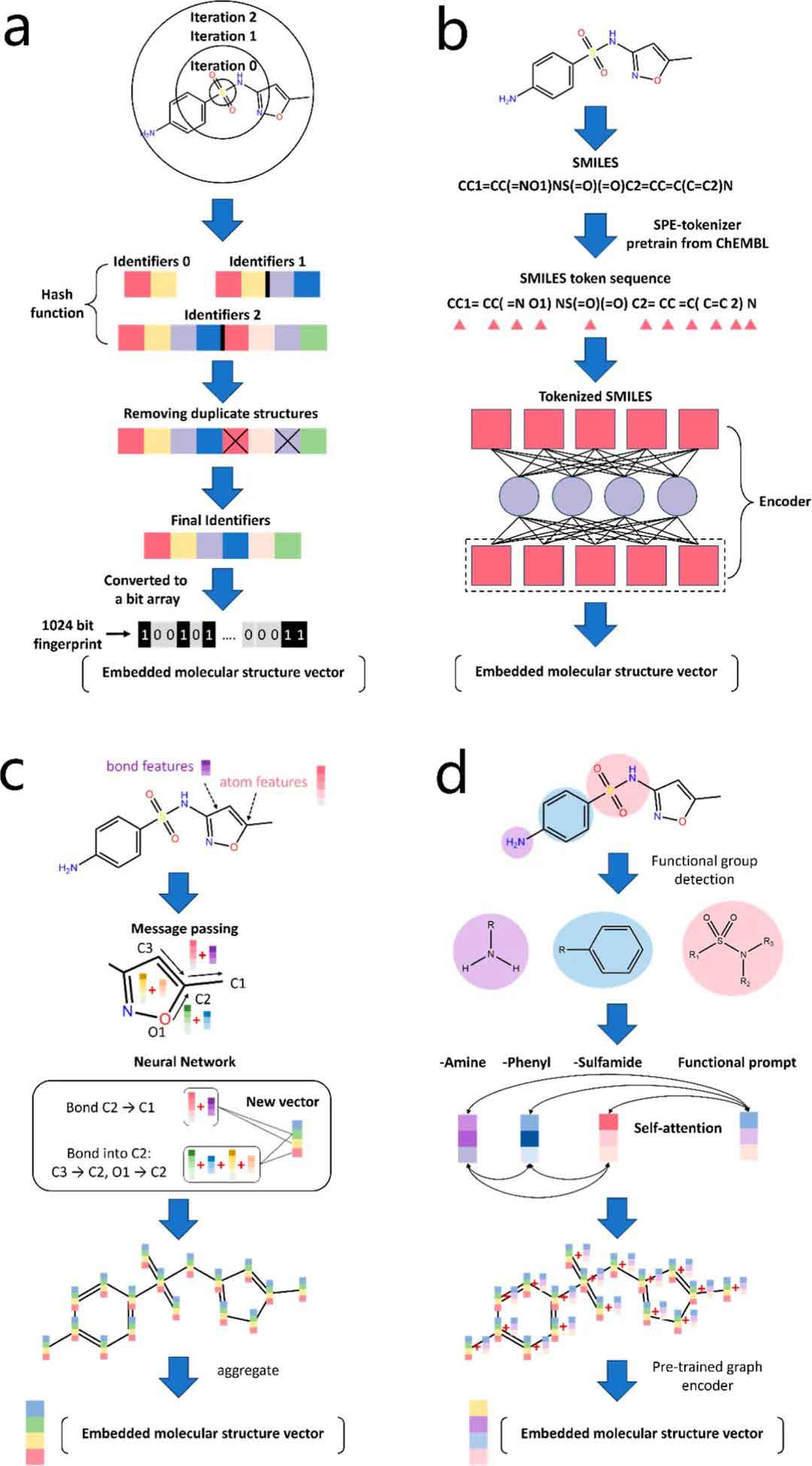
图1 利用(a) ECFP (b) SPE-MLP (c)D-MPNN和(d) KANO对分子结构嵌入途径进行比较可视化
本研究对ECs浓度特征(图2a)、水体理化性质(图2b)及臭氧处理条件(图2c)进行了统计分析。原水中ECs浓度变化范围大,处理后普遍降低。REC值分布均匀,部分超过1,可能源于臭氧反应迟缓或检测误差。为模拟真实饮用水环境,实验调控水质与处理条件,使其贴近常规工况。通过t-SNE算法降维分析,高维特征得以保留本征结构。图2d显示,喹诺酮、农药和PFAS聚类紧密,表现出类似特性与臭氧响应;而抗生素与药品分布较散,差异显著。进一步分析表明,PFAS与农药响应性差,REC偏高;喹诺酮类等则展现出良好反应性,验证机器学习在复杂结构预测中的有效性。
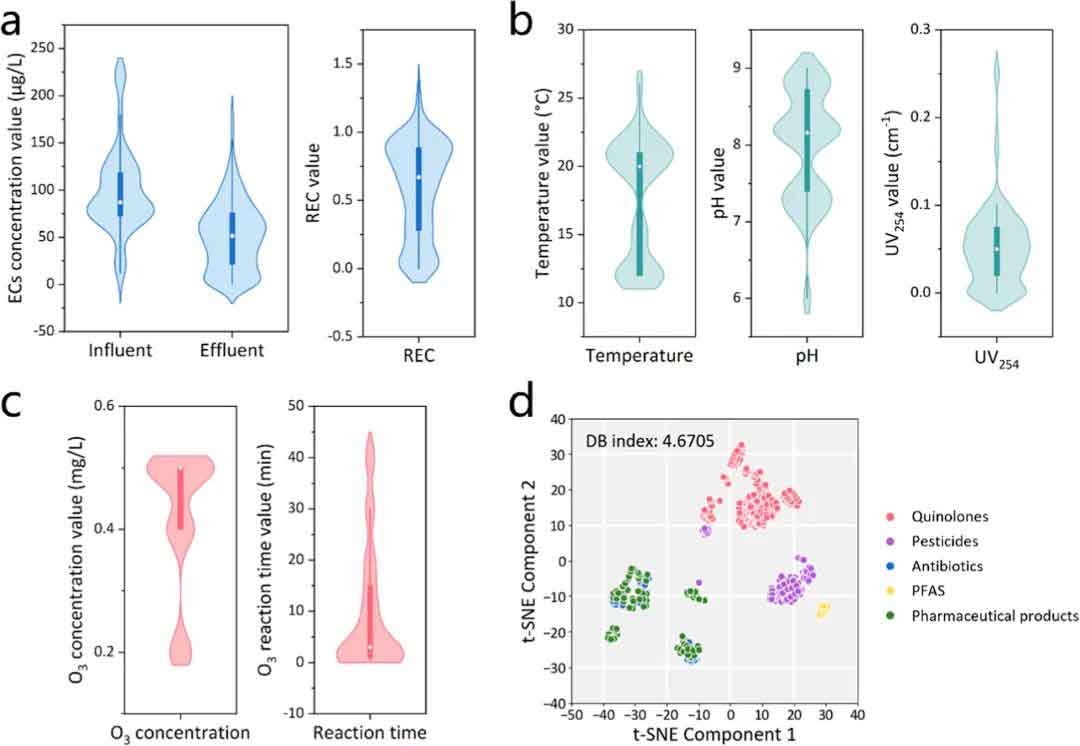
图2 新兴污染物数据特征分布与t-SNE聚类揭示模型代表性
对比分析显示,四种模型在验证集上均具备较高预测能力,DMPNN–EC表现最佳(R²=0.9717),KANO–EC次之,SPE–MLP–EC亦优于ECFP–EC(图3a–d)。在结构平衡划分测试集上,模型性能普遍下降(图3e),尤以SPE–MLP–EC退化最显著,因SMILES分词器难以处理新结构中的未知符号。ECFP–EC因指纹固定,难泛化新结构。相比之下,DMPNN–EC与KANO–EC依托图结构建模,可有效捕捉复杂结构特征,展现出较强的泛化能力,适用于多样性更高的环境污染物预测任务。
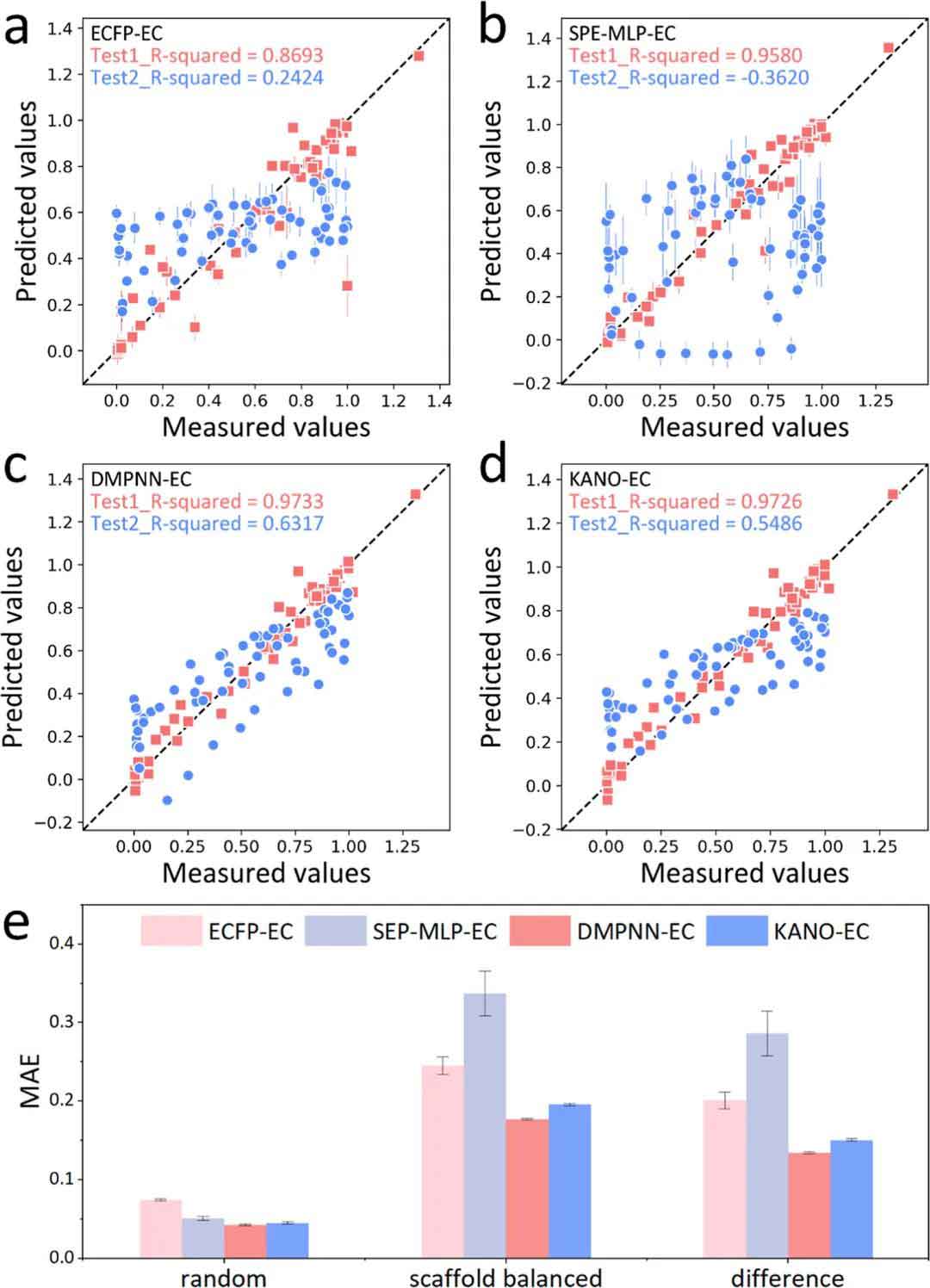
图 3 四种分子嵌入模型在随机与结构平衡数据集上的REC预测性能对比
为评估特征对模型预测能力的影响,研究对四种前馈神经网络(FNN)模型逐一剔除各类输入变量并比较R²变化(图4a)。结果显示,去除SMILES变量导致R²下降最显著,表明分子结构信息对REC预测至关重要。臭氧反应时间、UV254、臭氧浓度与温度的缺失也显著降低模型性能,说明这些环境因子在臭氧氧化中同样关键。相比之下,初始浓度(Cinitial)与pH对预测影响不显著,因实际水体pH波动较小。进一步计算模型平均R²差异,明确各变量的重要性排序为:SMILES > 时间 > UV254 > 臭氧 > 温度 > Cinitial > pH(图4b)。
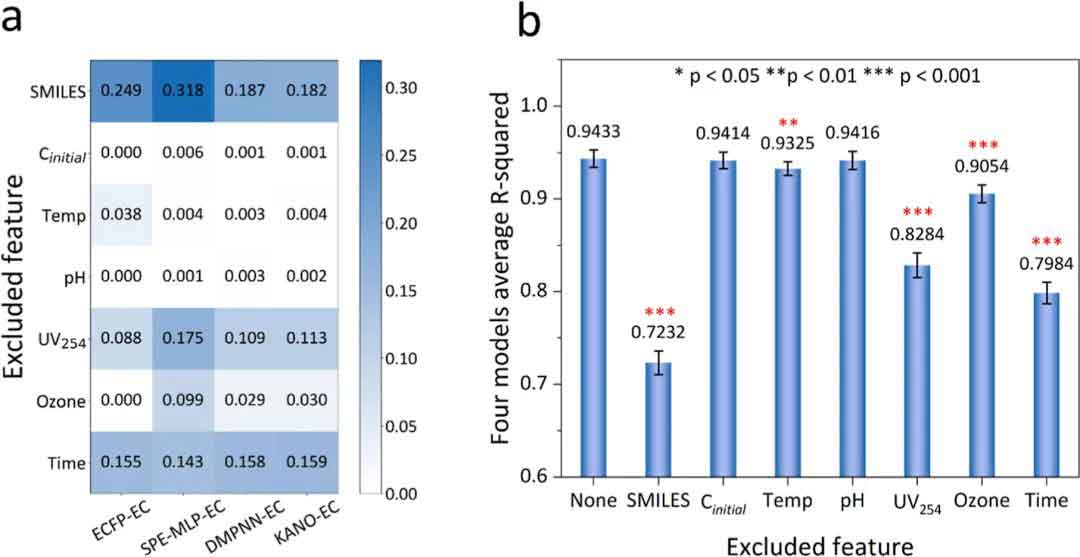
图 4 特征剔除下模型性能变化揭示多因素对REC预测的重要性排序
为识别对REC预测贡献最大的关键结构,图5展示了五类ECs代表分子的功能团注意力权重可视化。喹诺酮类的恩诺沙星中,羰基权重最高,其次为羟基、苯环和氟原子,符合Fukui函数分析所揭示的主要臭氧攻击位点(图5a)。除喹诺酮外,农药阿特拉津的三嗪环较稳定,甲基虽非活性位点,却表现出负向反馈(图5b)。抗生素磺胺二甲嘧啶显示苯环、伯胺和磺胺基为关键结构,匹配其药理骨架(图5c)。PFAS代表物PFNA因C–F键极强,几乎不被臭氧氧化降解(图5d)。药物卡马西平中,C=C双键为最主要反应位点,辅以苯环与羰基(图5e)。KANO–EC模型准确捕捉了功能团的结构–活性关联,为反应机制解析提供结构依据。
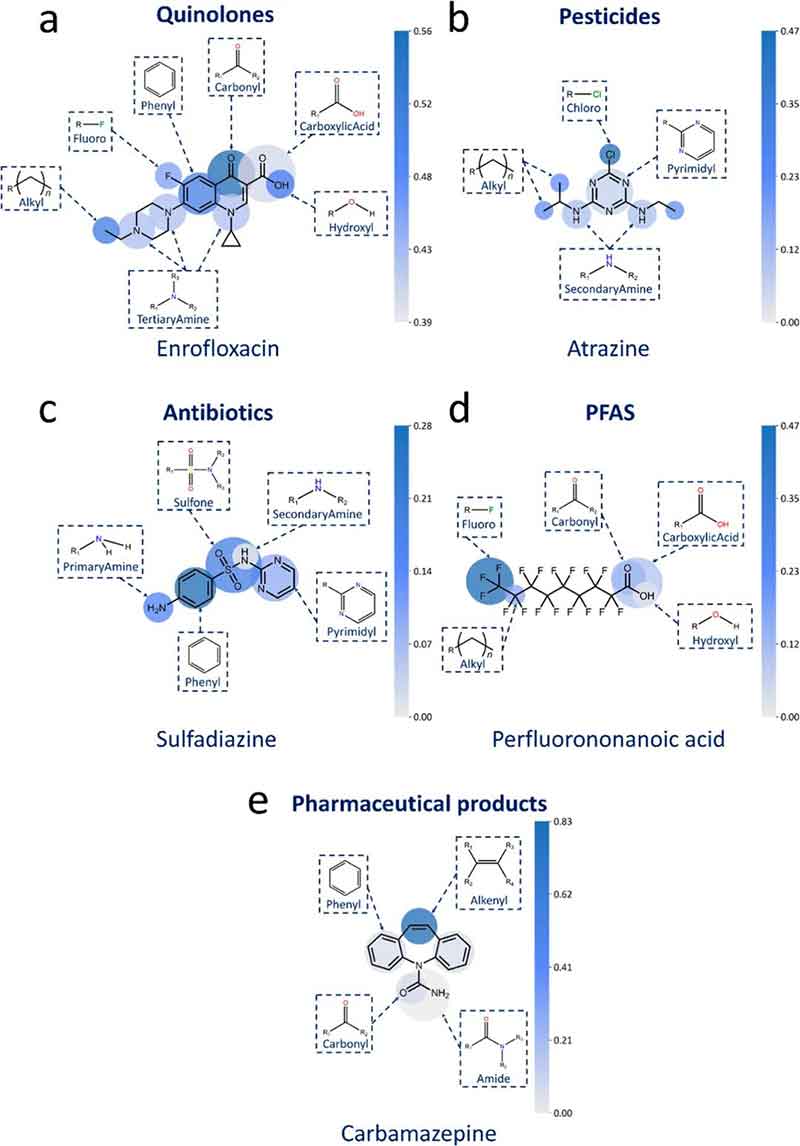
图 5 不同类别污染物中关键功能团的注意力权重可视化分析
图6聚焦于大环内酯类抗生素的结构-反应特征,涵盖克拉霉素(CLR)、红霉素(ETM)与替乐菌素(TLS)。结果显示,TLS具有更高的表观反应速率常数(图6a),主要因其C=C双键与醛基获得更高注意力权重(图6b)。三者均含羰基、酯、醚、叔胺和羟基等共通功能团。密度泛函理论计算表明,CLR中第5位氧原子与第7位碳原子具有显著的自由基Fukui函数,均属于羰基结构(图6c,d)。结合模型注意力权重,羰基被识别为主要反应位点。当缺乏显著亲电基团(如C=C或醛基)时,羟基自由基成为进攻羰基的主要反应机制。
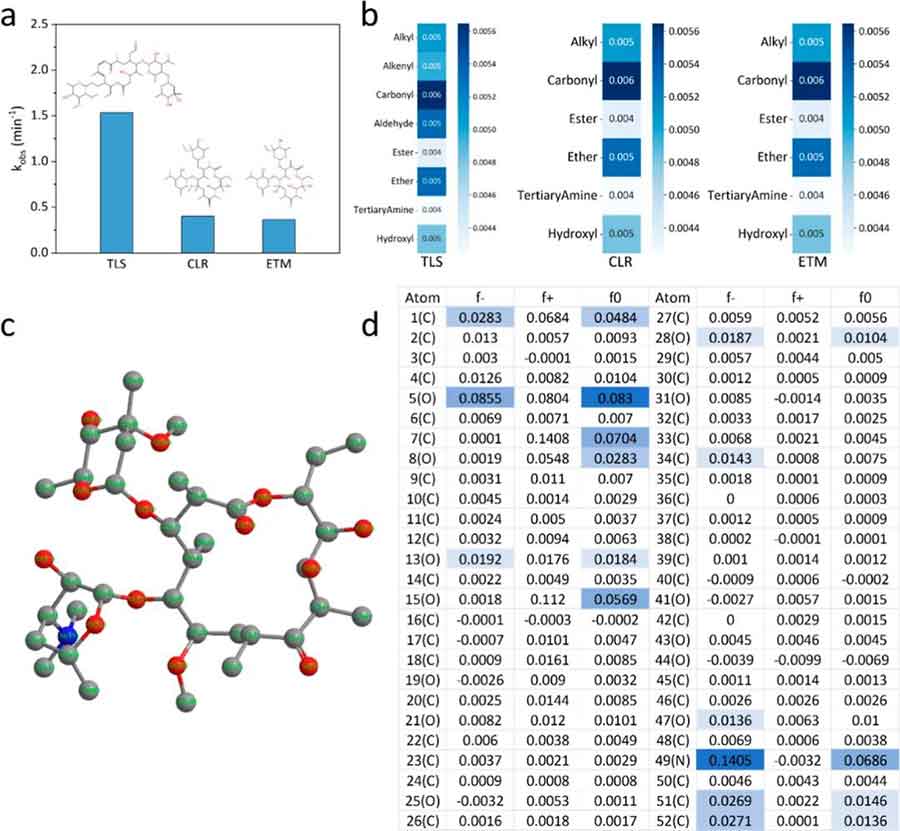
图 6 大环内酯类抗生素臭氧反应速率与关键结构位点的建模识别
主要结论本研究构建的融合分子结构嵌入的机器学习框架在预测新兴污染物残留率方面取得了显著成果,为大数据条件下的水处理智能控制及环境风险评估提供了新路径。该模型核心算法具备良好迁移性,可拓展应用于混凝、沉淀、活性炭吸附、消毒等多种水处理单元过程。通过将各处理环节模块化并集成为统一体系,可实现全过程优化控制,提升ECs在多阶段处理过程中的协同去除效率,为构建智能化、高效能的水处理系统奠定技术基础。
摘自:Machine Learning-Assisted Molecular Structure Embedding for Accurate Prediction of Emerging Contaminant Removal by Ozonation Oxidation
第一作者:Jiapeng Yue通讯作者:胡承志通讯单位:中国科学院生态环境研究中心、中国科学院大学原文链接:https://pubs.acs.org/doi/10.1021/acs.est.4c14193



 当前位置:
当前位置: 
 摘要
摘要




 上一篇:
上一篇: 返回列表
返回列表
